在基因组功能注释与个体差异研究中,SNP(单核苷酸多态性)和启动子区域是两个常被聚焦的核心内容。SNP能够揭示基因的遗传变异,是个体差异、疾病易感性等研究的重要基础;而启动子区则控制基因的表达,是调控研究的关键对象。DNASTAR作为经典的生物信息分析平台,提供了完整的SNP检测流程和启动子区域序列的挖掘与分析能力。围绕DNASTAR怎么进行SNP分析,DNASTAR怎么分析启动子区这个问题,本文将从操作步骤、关键参数设置和实际应用三个层面,详尽剖析这两个功能的实现方式,并扩展其在基因功能研究中的具体应用。

一、DNASTAR怎么进行SNP分析
在DNASTAR软件中,SNP分析主要依赖SeqMan NGen与SeqMan Pro两个模块进行原始数据比对、突变检测与结果展示。这个流程不仅适用于全基因组测序数据,也支持靶向区域或转录组数据的变异筛选。
1、建立SNP分析项目
打开SeqMan NGen,选择“Template Assembly”模式,导入参考基因组(FASTA格式)并加载样本测序数据(FASTQ文件)。软件支持多样本批量导入,并自动检测数据类型(单端/双端)。配置参数后点击“Run”开始比对和组装。
2、SNP自动识别与比对结果处理
比对完成后打开SeqMan Pro,载入.sqd项目文件。软件会自动识别所有碱基变异,包括SNP和InDel。通过“Variation Table”界面可查看突变位置、突变类型(转换/颠换)、突变频率、覆盖深度、质量得分等指标。
3、设置变异筛选参数
点击“Filter”按钮,用户可以设置筛选标准:
Minimum Depth(最小覆盖度,例如10×);
Minimum Variant Frequency(最小突变比例,一般设为20%);
Base Quality Score(最小碱基质量分);
筛选后的结果会实时更新,以确保只保留可信SNP位点。
4、注释与功能区定位
DNASTAR支持加载.gbk或.gff格式的注释文件,软件会自动识别每个SNP所在的功能区(如CDS、5’UTR、3’UTR、Intron等)。对处于CDS区域的SNP,还可判断其是否为同义或非同义突变。
5、SNP导出与数据共享
用户可通过“File>Export Variants”导出筛选后的SNP列表,格式为.csv或.txt,包含位置信息、参考碱基、突变碱基、样本信息、注释内容等字段。导出的表格可用于绘图、统计分析或交叉注释。
二、DNASTAR怎么分析启动子区
启动子区域是转录调控的重要元件,通常定义为TSS(转录起始位点)上游2kb至+100bp之间的DNA序列。通过DNASTAR的SeqBuilder Pro、GeneQuest和MegAlign Pro等模块,可以完成启动子序列提取、跨物种比对和保守元件搜索。
1、提取启动子区域序列
在SeqBuilder Pro中打开目标基因组序列并加载对应的基因注释(.gb/.gff格式),浏览至目标基因的转录起始位点(TSS)。点击基因名右键选择“Extract Upstream Region”,设置范围为-2000到+100,即可导出完整启动子区序列并保存为FASTA格式。
2、跨物种或多拷贝启动子比对
使用MegAlign Pro导入多个启动子序列(同一基因不同物种或同一物种不同样本),选择ClustalW、MAFFT或Muscle等算法进行多序列比对,识别高度保守的区域。可观察这些区域是否对应转录因子结合位点、TATA-box或CAAT-box等常规基序。
3、基序搜索与调控元件定位
打开GeneQuest,加载启动子区FASTA序列,选择“Motif Search”功能。可输入自定义的转录因子识别序列(如“TATAAA”)、结合位点模型或常见调控序列,软件会自动在正义链和反义链中搜索匹配区域,并标出位置、匹配得分与方向性。
4、启动子区域变异分析(结合SNP)
若在启动子区识别到SNP位点,可以进一步分析这些变异是否落在关键调控序列内。将SNP信息导入GeneQuest或SeqBuilder,并叠加在启动子结构上查看其对Motif区域的影响。例如某SNP可能破坏TATA-box,导致启动效率下降。
5、图形化展示与结果导出
在GenVision Pro中可加载启动子结构、转录因子Motif标记、SNP位置、保守区块等图层,生成清晰的结构图或调控图。图像支持导出为SVG、TIFF等格式,适合用于课题汇报或科研论文配图。
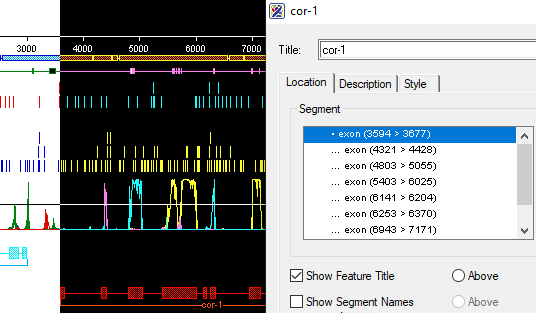
三、DNASTAR在变异与调控研究中的实际应用建议
除了SNP和启动子区的单独分析,DNASTAR工具组合还适用于更深入的功能注释和基因表达调控研究。以下是几个常见拓展应用方向,提升分析的科研价值。
1、联合SNP与表达数据进行eQTL筛选
通过DNASTAR输出的SNP列表与外部转录组表达量结合,可以开展表达数量性状位点(eQTL)关联分析。特别是启动子区的SNP可能对应显著表达差异,从而揭示调控机制。
2、建立样本间的调控差异比较体系
对于多个样本的启动子序列或比对数据,可通过MegAlign比对、保守性打分等手段,构建样本间的启动子多样性评分体系,进一步寻找可能的调控“热点”区域或保守块。
3、制作调控图谱数据库
结合DNASTAR的图形化能力和批量输出功能,研究者可整理全基因组启动子区域的结构图、Motif分布和变异图谱,作为研究组内部的调控结构资源。
总结
DNASTAR怎么进行SNP分析,DNASTAR怎么分析启动子区不仅是基础的功能模块问题,更是基因组注释与功能分析之间的桥梁。通过灵活使用SeqMan、GeneQuest、SeqBuilder和GenVision等模块,用户可以从原始数据入手,构建起完整的变异检测、调控结构分析、图谱展示的科研链条,在分子生物研究中实现更具深度的发现与表达。

