在基因组学和分子生物学研究中,序列比对不仅是分析变异、注释功能的重要基础,也直接关系到后续的结构建模与表达验证。特别是在面对大规模、多样化的序列数据时,比对的布局策略、算法选择和性能调优就显得尤为关键。DNASTAR作为一款专业的生物信息软件,其在比对布局优化和长序列处理方面具备较为成熟的解决方案。本文围绕DNASTAR如何优化比对布局DNASTAR如何比对长序列这两个维度展开,帮助科研人员提升比对的效率与准确性。
一、DNASTAR如何优化比对布局
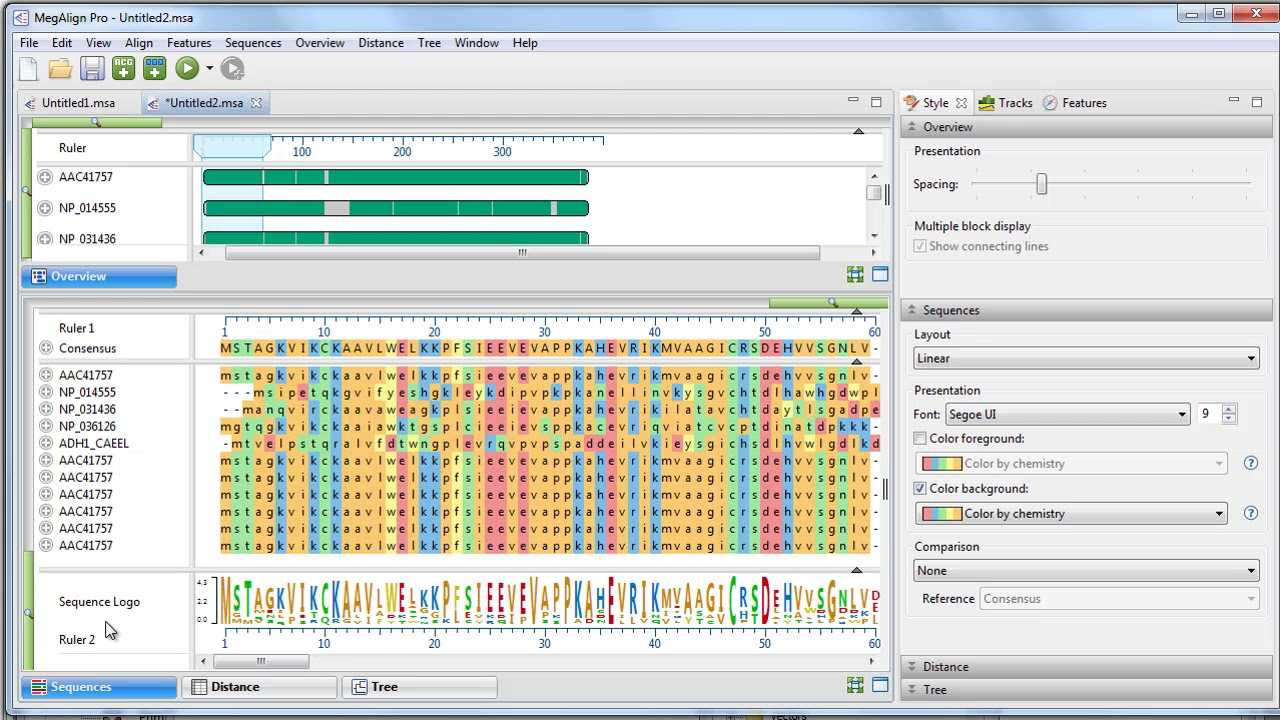
在进行多序列比对时,序列的可视化布局直接影响分析的直观性与比对质量的判断。DNASTAR主要通过MegAlign模块来处理多序列比对,并支持多种比对算法和布局方式。
1.选择合适的比对算法
DNASTAR提供多种比对算法:ClustalW、MAFFT、MUSCLE、JotunHein等。其中:
ClustalW:适用于保守区域突出的多序列比对。
MUSCLE:速度快、适合中等数量序列。
MAFFT:对变异频繁序列比对效果佳。
JotunHein:适用于高复杂度序列(如含大片段插入)。
根据样本特征选择算法,是优化比对布局的第一步。
2.调整序列顺序与对齐方式
DNASTAR允许用户手动拖动序列顺序,也可以按相似性自动排序,增强比对的对比性。在“Layout”设置中,可以选择横向排列(默认)、纵向展开或树状结构布局,便于观察片段错配或热点区域。
3.可视化参数调整
比对结果支持颜色编码显示保守性、差异性、Gap区域等。用户可选择以下显示策略:
Identityshading:显示相同碱基为同色,差异高亮。
Conservationshading:根据保守性深浅标色。
Mismatchhighlighting:突显突变位点与变异类型。
4.区域折叠与扩展功能
面对较长序列,MegAlign支持折叠中间无差异区域,仅显示突变、Gap等有效区段。这一功能可在“CollapseConservedRegions”选项中启用,极大提升可读性。
5.动态比对刷新机制
修改序列后,无需重新载入,DNASTAR支持实时刷新比对结果。对于突变编辑、多版本修订等操作尤为实用。
6.导出与注释同步
比对完成后,用户可将结果导出为PDF、FASTA对齐格式,并选择是否包含注释与颜色标记。此外,比对区域可直接同步到SeqBuilder中进行进一步功能分析,如ORF预测、引物设计等。
二、DNASTAR如何比对长序列
比对大于10kb甚至上百kb的长序列(如基因组片段、病毒全长、转录本拼接体)时,常规的比对策略可能出现内存瓶颈、比对失真或过度裁剪的问题。DNASTAR针对长序列处理做了多项优化。
1.启用专用长序列比对算法
在MegAlignPro中可启用“Gene-to-Gene”比对模式,该模式基于改进的动态规划算法,能够处理上百kb长度的DNA序列对齐,同时保留大段结构特征。
2.分段比对处理
对于超大序列,DNASTAR建议使用“分段比对”策略。可将全长序列按基因、Exon、功能模块等拆分为若干子序列,再分批进行比对。此法可规避一次性加载带来的内存压力。
3.使用GenVisionPro整合长序列结果
比对完成后,若需要将多段比对拼接为完整图谱,DNASTAR支持导入至GenVision模块,生成基因组图谱、结构域分布图等,实现结构-功能联合分析。
4.长序列Gap管理机制
在长序列中,大量的Gap区域会影响比对可读性与统计结果。MegAlign允许用户设定GapPenalization策略,比如设置“GapOpenPenalty”和“GapExtensionPenalty”,以控制插入/缺失比对权重,从而在长序列中减少无意义比对。
5.多线程加速与缓存设置
DNASTAR支持多核并行加速计算,建议在设置中开启“UsemultipleCPUs”,并将缓存路径设定为SSD磁盘,有效提升大数据处理效率。
6.导出长序列比对结果的兼容格式
MegAlign支持将长序列比对导出为:
多FASTA格式(AlignedFASTA)
Nexus文件(供BEAST等进化分析使用)
GFF注释对齐格式
比对图像(高分辨率SVG或TIFF)
便于在下游软件(如MEGA、RaxML)中继续分析。
三、如何在DNASTAR中同步结构域与比对结果
为了更高效地利用比对信息,DNASTAR还允许用户将蛋白结构域信息叠加到比对视图中,实现结构-比对联动。
1.导入蛋白结构域注释
若已知序列对应蛋白结构域(如Pfam、InterPro结果),可通过“ImportAnnotation”导入GFF或TXT格式的域注释文件。
2.显示结构域与突变重叠区域
在比对视图中,DNASTAR会将结构域高亮显示,并标出其中的SNP或Indel位点,便于发现潜在的功能改变区域。
3.结合Protean模块分析结构影响
用户可将该区域同步发送至Protean模块,预测突变是否导致二级结构改变、疏水性变异或蛋白稳定性下降。
4.构建保守性图谱
MegAlign还支持根据比对保守性生成“ConsensusGraph”,可视化每个位点的保守程度。这对于识别关键功能残基非常有效。
结语
综上所述,针对DNASTAR如何优化比对布局DNASTAR如何比对长序列这两个问题,DNASTAR提供了从算法、显示、性能到数据导出的全流程解决方案。无论是简单的多序列比对,还是复杂的长序列整合,DNASTAR都能通过模块联动、可视化增强以及智能参数调节,帮助用户获得高质量的比对结果。这对于结构域注释、变异分析、功能预测等生物信息任务而言,是一个不可或缺的高效平台。

