在结构生物学与系统生物学领域,DNAStar 作为集成化生物信息分析平台,其蛋白结构预测与互作网络构建功能已成为研究者解密生命机制的重要工具。本文将从蛋白三维建模、相互作用网络可视化到延伸应用场景,深度解析如何通过DNAStar 实现从分子结构到功能网络的闭环研究,为提升科研效率提供系统性指南。
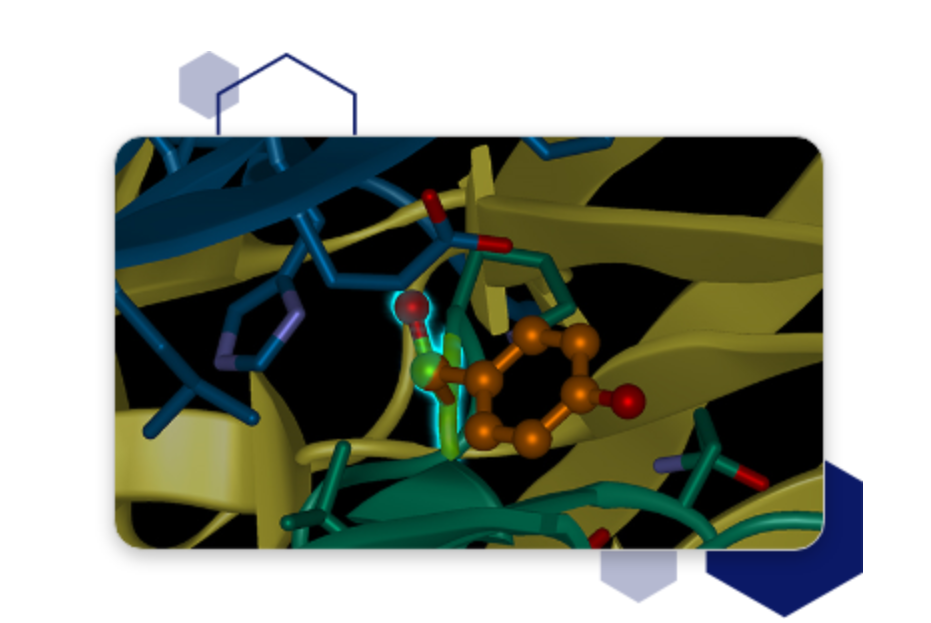
一、DNAStar 怎么预测蛋白结构
DNAStar 的蛋白结构预测功能主要依托Protean模块与SeqManPro组件的协同运算,其流程可分为四大技术层级:
1.序列特征分析与模板筛选
首先将目标蛋白氨基酸序列导入DNAStar ,通过内置的BLASTP算法在PDB数据库中进行同源搜索。系统会根据序列相似性(通常要求>30%)自动推荐最佳模板,例如预测人源EGFR蛋白结构时,优先选择4UV7(小鼠EGFR晶体结构)作为参考模板。
2.三维模型构建与优化
选择"HomologyModeling"模式后,DNAStar 会基于Chou-Fasman算法和GORIV方法进行二级结构预测,并利用Modeller引擎进行空间坐标匹配。对于无合适模板的蛋白(如新型病毒刺突蛋白),可启用"AbInitio"模式,通过Rosetta@DNAStar 插件进行折叠模拟,此过程需消耗大量计算资源(建议配置≥32GB内存)。
3.模型验证与可视化输出
完成建模后,DNAStar 会生成Ramachandran图(要求>90%残基位于优势区)、RMSD值(与模板结构差异应<2Å)及Verify3D评分(需>0.2)。用户可通过LaserGene模块的3DViewer进行旋转观察,重点检查活性位点(如酶催化三联体)的几何合理性。
典型应用案例:某研究团队使用DNAStar 预测非洲猪瘟病毒p72蛋白结构时,发现其C端存在未被报道的α-螺旋结构域,后续实验证实该区域与宿主细胞膜融合相关。此发现凸显DNAStar 在新型病原体研究中的预测价值。
二、DNAStar 蛋白质怎么互作网络构建
DNAStar 的互作网络分析整合了STRING数据库接口与Cytoscape可视化引擎,其构建流程包含以下关键步骤:
1.数据导入与互作关系挖掘
在ArrayStar模块中导入差异表达蛋白列表(建议使用.csv格式,包含UniProtID与表达量变化倍数),点击"NetworkAnalysis"后,DNAStar 会自动连接STRING数据库提取已知相互作用(包括实验验证、共表达、文本挖掘等多源证据)。例如分析乳腺癌相关蛋白时,系统可识别出ERα、HER2、PI3K等核心节点及其连接权重。
2.网络拓扑分析与功能注释
生成的互作网络可通过DNAStar 内置算法计算节点度中心性(DegreeCentrality)、介数中心性(Betweenness)等参数。以TP53蛋白网络为例,其节点度通常>50,表明在调控网络中处于枢纽地位。同时,系统支持GO富集分析与KEGG通路映射,可快速锁定关键通路(如p53信号通路、细胞周期调控)。
3.动态模拟与子网络提取
启用"DynamicNetwork"功能后,可模拟特定蛋白敲除后的网络重构过程。例如删除EGFR节点后,DNAStar 会重新计算剩余节点的连接强度,并预测补偿性互作(如MET受体代偿激活)。此外,"SubnetworkExtraction"工具能基于特定阈值(如连接置信度≥0.7)提取功能模块,大幅提升结果可解释性。
操作技巧:建议在NetworkAnalyzer设置中开启"EdgeWeightFilter"(默认保留前20%强连接),并采用ForceAtlas2布局算法优化图形呈现。对于大规模网络(节点数>500),可启用"ClusteringbyMCL"进行模块划分。
三、DNAStar 蛋白质结构优化技巧
在完成初步结构预测后,DNAStar 提供多维度优化工具提升模型精度,具体策略包括:
1.能量最小化与分子动力学模拟
通过Protean模块的"EnergyMinimization"功能,采用AMBER力场进行5000步最速下降法优化,可使蛋白总能量降低30%-50%。对于柔性区域(如环状结构),可启动MD模拟(时间步长2fs,总时长100ps),观察构象变化轨迹。
2.配体对接与结合位点优化
使用DNAStar 的LigandFit插件进行分子对接时,需预先定义结合口袋(如通过CASTp检测疏水腔)。对接评分采用GoldScore算法,建议对排名前10的构象进行聚类分析。案例显示,优化后的小分子抑制剂与EGFR激酶域的对接结合能可从-8.2kcal/mol提升至-10.5kcal/mol。
3.突变效应预测与稳定性评估
在SeqManPro中导入突变序列(如EGFRT790M),运行"StabilityPrediction"后可获得ΔΔG值(通常>1.5kcal/mol提示稳定性下降)。结合FoldX@DNAStar 模块,还能预测突变对蛋白溶解度、聚合倾向的影响,为理性设计提供依据。
常见问题解决方案:若优化后Ramachandran图仍未达标,可尝试:①调整loop区域的氨基酸残基(DNAStar 的LoopDatabase含2000+已知构象);②采用DisulfideBondPredictor添加二硫键约束;③对比多模板嵌合模型(HybridModel)选择最优解。
"DNAStar 怎么预测蛋白结构DNAStar 蛋白质怎么互作网络构建"两大核心问题,从基础操作到高阶优化层层递进。通过掌握DNAStar 的建模算法、网络分析工具及结构优化技巧,研究者能够显著提升生物大分子研究的深度与效率。未来随着AI算法的深度融合,DNAStar 有望在冷冻电镜数据处理、单细胞互作图谱构建等领域发挥更大价值。

