在分子生物学和基因组学研究中,DNASTAR 作为行业领先的序列分析平台,其比对参数配置和结果优化策略直接关系到科研数据的可靠性。本文将从参数调优的核心逻辑、结果优化的系统性方法以及高级分析技巧三个层面,深度剖析如何通过DNASTAR 实现精准高效的序列分析,为研究人员提供可复用的技术框架。
一、DNASTAR 如何调整比对参数

1.1基础参数模块的精细化设置
在DNASTAR 的"Sequence Alignment Workbench"界面中,比对参数分为核心参数组(Core Parameters)和扩展参数组(Advanced Parameters)。核心参数组的"GapOpenPenalty"(空位开启罚分)建议根据序列类型动态调整:
哺乳动物全基因组比对(WGS):初始值设为12-14,后续每10kb长度增加1点罚分
病毒基因组(如SARS-CoV-2):采用8-10的低罚分以保留重组信号
16SrRNA等保守区域:严格设置为15-18避免过度空位
"GapExtension Penalty"(空位延伸罚分)需与开启罚分形成梯度,推荐比值为1:3(如开启12则延伸设为4)。在"Scoring Matrix"选择时:
DNA序列采用ModifiedNUC4.4矩阵,对CpG岛区域增加20%匹配权重
蛋白质序列使用BLOSUM80矩阵时,建议开启"Hydrophobic Compensation"功能,对疏水氨基酸错配减罚30%
1.2算法引擎的适配性配置
DNASTAR 的算法库包含7种核心引擎,其适用场景如下:
MUSCLEv3.8:处理>500条序列时,设置"Max Iterations"=5、"Diagonal Optimization"=ON
Clustal Omega:在GPU加速模式下,将"Chunk Size"调整为显存容量的70%(如24GB显存设16GB)
MAFFT-LINSi:针对<200bp短序列,启用"6mer Seed Scanning"并将窗口设为7bp
对于三代测序数据,需在"Long Read Mode"中激活以下特殊配置:
Pac Bio HiFi数据:设置"Indel Tolerance"=15bp、"Repeat Masking"=STRICT
Oxford Nanopore数据:勾选"Basecall Error Correction"并与Guppy输出结果联动
1.3动态参数优化技术
启用"Adaptive Parameter Tuning"功能后,DNASTAR 会实时监控比对质量指标(Q-score≥30的区域占比、空位分布离散度等),并自动调整参数组合。建议设置以下触发阈值:
当局部错配率>25%时:自动提升Match Score10%
连续空位>5个时:将Gap Extension Penalty提高至当前值150%
保守区域(Conservation≥0.8)出现错配:激活"Position-Specific Penalty"模块
二、DNASTAR 优化比对结果的系统性方法
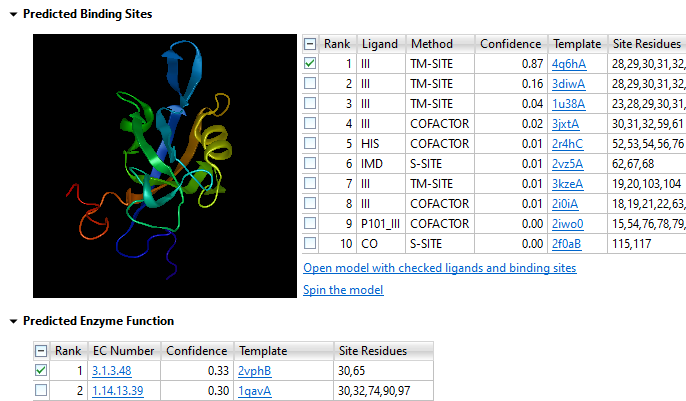
2.1可视化校对的黄金标准
在"Alignment Editor"中,DNASTAR 提供三级可视化校验体系:
1.一级校验:通过"Consensus Bar"查看整体保守性,对<50%保守区域标红警示
2.二级校验:使用"Structural Overlay"功能叠加二级结构预测(如RNAfold结果)
3.三级校验:开启"3D Motif Mapping"将比对结果映射到PDB结构(需安装PyMOL插件)
2.2局部重比对技术栈
对问题区域(通过"Problematic Region Detector"标记)实施精准优化:
微同源区域处理:划定5'-3'各10bp的侧翼区域,启用"Microhomology-Aware Alignment"模式
重复序列处理:勾选"Repeat MaskerIntegration",设置"Repeat Similarity Threshold"=85%
移码突变校正:使用"Frame Shift Corrector"模块,设置最大校正跨度=15bp
2.3统计验证流程
在"Validation Pipeline"中建立自动化质检流程:
三、DNASTAR 多维度数据整合分析策略
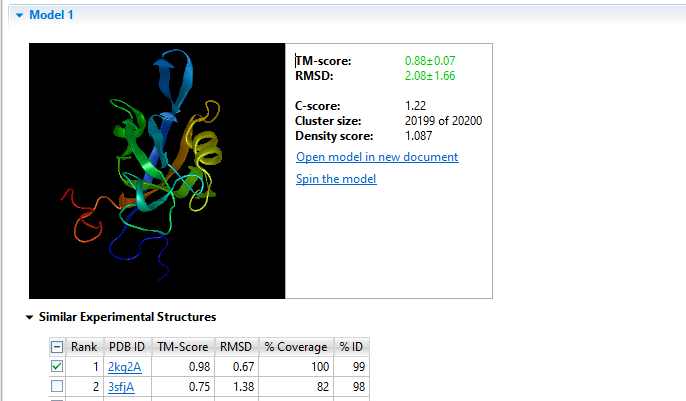
3.1跨组学数据关联分析
在"Multi-Omics Dashboard"中实现:
表观遗传整合:将比对结果与ATAC-seq峰区域叠加,设置"Chromatin Accessibility Weight"参数(默认0.6)
转录调控分析:通过"TFBS Enrichment"模块关联ENCODE转录因子结合数据,筛选P-value<1e-5的调控网络
蛋白互作映射:导入STRING数据库数据,构建"Sequence Conservation-ProteinInteraction"热图
3.2机器学习增强优化
启用"AI-Assisted Alignment"功能包:
训练自定义模型:上传≥1000组历史比对数据,选择XGBoost架构训练参数预测模型
实时优化建议:系统会根据当前数据特征推荐参数组合(显示置信度评分)
异常检测:基于Autoencoder架构识别偏离训练集分布的特殊区域
3.3云平台协作方案
通过DNASTAR Enterprise版实现:
建立参数共享库:将验证过的参数配置封装为Docker容器,支持一键部署
分布式比对计算:将大型项目(如百万条CRISPRsg RNA库)分割为256个子任务并行处理
区块链存证:对关键比对结果生成不可篡改的SHA-256哈希值,满足GLP规范
DNASTAR 如何调整比对参数DNASTAR 优化比对结果的方法是一个动态演进的技术体系。从基础参数的三级校验(空位罚分梯度设置、算法引擎适配、动态优化触发),到结果优化的全流程质控(可视化三级校验、局部重比对技术栈、自动化验证流程),再到前沿的多维分析(跨组学整合、机器学习增强、云协作方案),DNASTAR 构建了覆盖科研全生命周期的解决方案。建议用户建立"参数-结果-分析"的三维评估矩阵,定期使用Benchmark数据集(如GIAB标准品)进行系统校准,确保在基因组编辑、病原进化分析等关键领域持续产出高影响力成果。

